课程目录:
1、数据挖掘&概述
2、GEO数据库
3、功能分析
4、TCGA数据库挖掘
5、Oncomine数据库
一、数据库挖掘与概述
GEO数据库
芯片数据http://www.biowolf.cn/GEOchip/geo_search.html
TCGA数据库
癌症数据库,临床信息http://www.biowolf.cn/TCGA/tcga_combat.html
数据挖掘
二、GEO数据库
1、GEO在线工具
Analyze with GEO2R工具使用
GEO网址:http://www.ncbi.nlm.nih.gov/geo/
http://www.ncbi.nih.gov/geo/geo2r/
实例芯片代号:GSE79973
2、质量分析
3、数据处理
4、差异表达
三、功能分析
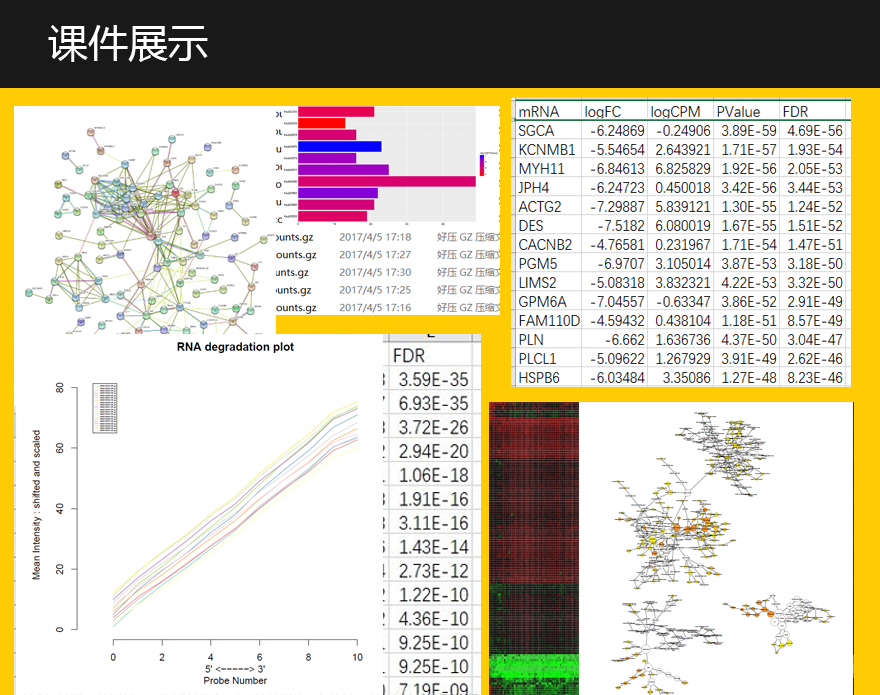
1、GO富集:GO(Gene ontology)按照生物途径(Biology Process),分子功能(Molecular Function)和细胞定位(Celluar Location)
对基因进行注释和分类。通过差异基因进行GO terms富集度统计学分析,计算出差异基因GO term的p-value和p-value的FDR值(q-value),定位差异基因最可能相关的GO term。
工具:1、DAVID;2、BINGO
2、KEGG富集分析
R做柱状图
3、蛋白互作网络构建
String在线工具
网址:https://string-db.org/
四、TCGA数据库挖掘
1、数据下载
gdc工具下载转录本数据
2、差异分析
edgeR包做差异分析
火山图、热图
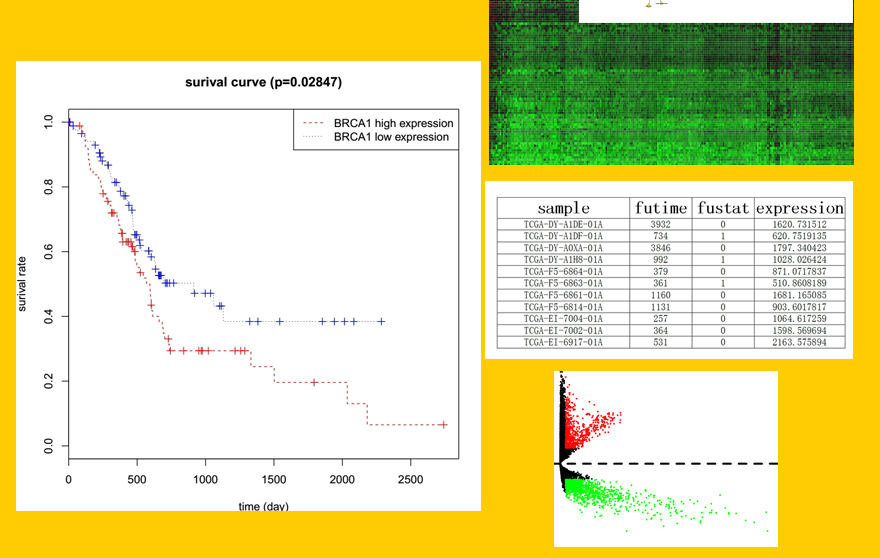
3、生存分析
5年生存曲线绘制
五、Oncomine数据库分析
Oncomine是目前世界上最大的癌基因芯片数据库和整合数据挖掘平台,旨在挖掘癌症基因信息。到目前为止,该数据库已经收集了715个基因表达数据集,86733个癌症组织和正常组织的样本数据。Oncomine拥有最全的癌症突变谱、基因表达数据以及相关的临床信息,可利于发现新的生物标记物或新的治疗靶点。
Oncomine的使用并非免费的,广大高校的小伙伴们可要好好的珍惜自己的学生身份,在校生可以通过学校的邮箱注册使用。
通过oncomine,可以进行差异表达分析、共表达分析,查找某种癌症中差异表达的基因,确定目的基因,确定研究方向。对于研究方向还没有确定的临床医生,如果想要通过芯片筛选确定感兴趣的研究分子,可以通过oncomine数据挖掘的方法确定研究方向。不仅可以为您节省科研成本,而且其信息也更加全面。






 赣公网安备 36090202000081号
赣公网安备 36090202000081号


最有帮助的评价(2)
课程包含很多内容,而且提供课程教程,真心不错
课程讲了很多知识,一口气学习了3个数据库的分析,适合我这种没基础的,原来生信可以这样分析,老师解答也很好